Forschung
NCCR AntiResist revolutioniert die Antibiotikaforschung und -entwicklung durch die Entwicklung von Labormodellen, die die physiologischen Wechselwirkungen zwischen Bakterien und menschlichem Gewebe auf der Grundlage der in Patientenproben beobachteten Bedingungen nachbilden. Diese innovative In-vitro-Modellierung von In-vivo-Bedingungen bietet neue Möglichkeiten für die Antibiotikaforschung und gibt neue Hoffnung im Kampf gegen antibiotikaresistente Infektionen.
Scrollen Sie nach unten, um einen Überblick über unsere Forschungsarbeiten zu erhalten, oder klicken Sie direkt auf die unten stehenden Links.
Warum ist unsere Forschung notwendig?
Da sich Bakterien schnell weiterentwickeln, können sie Resistenzen gegen Antibiotika entwickeln. Die Fähigkeit von Bakterien, die Wirkung von Antibiotika abzulenken, ist zu einer globalen medizinischen Bedrohung geworden. Nach dreißig Jahren Stagnation in der Entwicklung neuer Antibiotika stehen wir vor der sehr nahen Aussicht auf eine Post-Antibiotika-Ära, die eine massive gesundheitliche und wirtschaftliche Belastung mit sich bringt.
Während neue, auf herkömmliche Weise entdeckte Antibiotika eine vorübergehende Atempause verschaffen, strebt AntiResist danach, nachhaltigere und globalere Ansätze zu entwickeln, um die Innovationslücke im Bereich der Antibiotika zu schließen. Mit einer grundlegenden Verlagerung hin zur In-vivo-Antibiotikaforschung (innerhalb des lebenden Organismus) schaffen wir die Grundlage für die kontinuierliche Entdeckung neuer Antibiotika, um mit der Resistenzentwicklung für kommende Generationen Schritt zu halten.

Was unterscheidet unsere Forschung von anderen?
Inwiefern sind herkömmliche Forschungsansätze begrenzt? Das innere Milieu des menschlichen Körpers ist physikalisch, chemisch und biologisch komplex und setzt Bakterien durch Ernährungsbeschränkungen oder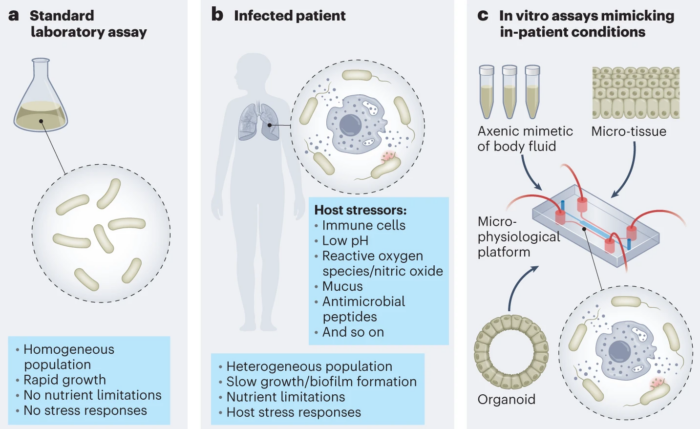 Abwehrmechanismen des Wirts verschiedenen Belastungen aus. Dieses hochgradig anpassungsfähige Wirtsmilieu wird durch verschiedene menschliche Zellen und Biomoleküle geprägt (b), die bei herkömmlichen Ansätzen zur Entwicklung von Antibiotika nicht berücksichtigt werden. Ein Beispiel dafür, dass eine Laborkultur den menschlichen Wirt nicht widerspiegelt, stammt aus der Genetik, wo bakterielle Gene identifiziert wurden, die in einer Laborkultur inaktiv sind, aber aktiv werden, wenn Bakterien menschliches Gewebe infizieren, und deren Wachstum unterstützen. Dies offenbart eine erhebliche Lücke in der Antibiotikaforschung, und es könnte noch viele weitere geben. Ohne ein genaues Verständnis der Wechselwirkungen zwischen Bakterien und dem menschlichen Körper fällt es Wissenschaftlern schwer, relevante Schwachstellen von Bakterien und mögliche Wirkmechanismen von Antibiotika zu identifizieren.
Abwehrmechanismen des Wirts verschiedenen Belastungen aus. Dieses hochgradig anpassungsfähige Wirtsmilieu wird durch verschiedene menschliche Zellen und Biomoleküle geprägt (b), die bei herkömmlichen Ansätzen zur Entwicklung von Antibiotika nicht berücksichtigt werden. Ein Beispiel dafür, dass eine Laborkultur den menschlichen Wirt nicht widerspiegelt, stammt aus der Genetik, wo bakterielle Gene identifiziert wurden, die in einer Laborkultur inaktiv sind, aber aktiv werden, wenn Bakterien menschliches Gewebe infizieren, und deren Wachstum unterstützen. Dies offenbart eine erhebliche Lücke in der Antibiotikaforschung, und es könnte noch viele weitere geben. Ohne ein genaues Verständnis der Wechselwirkungen zwischen Bakterien und dem menschlichen Körper fällt es Wissenschaftlern schwer, relevante Schwachstellen von Bakterien und mögliche Wirkmechanismen von Antibiotika zu identifizieren.
Wie können wir diese Lücke schließen? Die Identifizierung und Nutzung der Möglichkeiten, die die menschliche Wirtsumgebung bietet, steht im Mittelpunkt unseres Ansatzes bei NCCR AntiResist. Zunächst entnehmen wir Gewebeproben von infizierten Patienten und untersuchen diese genau. Anschließend replizieren wir diese In-vivo-Infektionsumgebung in vitro (im Labor), indem wir axenische, Mikorgewebe- und Organoidmodelle erstellen, um die Infektion und Behandlung zu untersuchen (c).
Die genaue Modellierung von Infektionsbedingungen bei stationären Patienten in vitro schafft eine realistische physiologische Umgebung, um In-vivo-Zustände zu untersuchen, die mit hochauflösender Mikroskopie und anderen Methoden beobachtet werden können, um die Dynamik des Infektionsprozesses sowie die Wirksamkeit von Antiinfektiva aufzuklären. Wir glauben, dass diese innovative Strategie zur Nachbildung der Umgebung des menschlichen Körpers die Infektionsforschung grundlegend verändern und dazu beitragen wird, die Infektionskontrolle für zukünftige Generationen sicherzustellen, indem sie die Pipeline für die Entdeckung von Antibiotika wiederbelebt.
Weitere Einzelheiten zu unserem Forschungsansatz finden Sie im Kommentar von Sollier et al. aus dem Jahr 2024 in Nature Microbiology.
Interdisziplinärer Ansatz
Das NCCR AntiResist-Forschungsteam umfasst Experten aus den Bereichen Mikrobiologie, Biochemie, Humanbiologie, Pharmakologie, Bioingenieurwesen und klinische Medizin aus verschiedenen Institutionen in der Schweiz und im Ausland, die gemeinsam an der Identifizierung neuer Antibiotika und antimikrobieller Strategien arbeiten.
Diese Vielfalt an Perspektiven und Fachkenntnissen ermöglicht es uns, schnell und entschlossen zusammenzuarbeiten, um die Forschung und Entwicklung im Bereich Antibiotika voranzutreiben und bisher vernachlässigte Möglichkeiten effektiv zu untersuchen und zu nutzen. Indem wir uns auf einige der gefährlichsten menschlichen Krankheitserreger konzentrieren, werden wir neue antimikrobielle Ziele und Schwachstellen von Bakterien identifizieren, Methoden und Technologien verbessern, die zur Überwindung der Hindernisse herkömmlicher Ansätze erforderlich sind, und eine neue Generation antimikrobieller Strategien entwickeln.
Zielbakterien
Unsere Forschung konzentriert sich auf Mikroben, die die größte Gefahr für die menschliche Gesundheit darstellen.
Escherichia coli, Pseudomonas aeruginosa und Staphylococcus aureus gehören zu den tödlichsten bakteriellen Krankheitserregern, die die globale Gesundheit bedrohen, und Brucella melitensis ist in mehreren Regionen weltweit ein bedeutendes und endemisches Problem. Unsere Forschung basiert auf einem tiefgreifenden Verständnis der Mechanismen, mit denen diese Krankheitserreger den derzeitigen Behandlungsmethoden entgehen. Unsere Mission ist es, Schwachstellen in ihrer Biologie zu identifizieren und diese für die Entwicklung neuartiger Therapien zu nutzen.
Unser Ansatz in Kürze
Unsere Forschung konzentriert sich auf drei Hauptziele.
Erstens wollen wir In-vitro-Modelle entwickeln, die wichtige Aspekte der Infektion und Behandlung bei Patienten nachbilden. Diese Modelle werden mit gesundem menschlichem Gewebe/Blut und klinischen Proben, die mit einem der vier Schwerpunktpathogene infiziert sind, verglichen und mit klinisch relevanten Isolaten und verfügbaren Daten für Standardantibiotika validiert. Wir verbessern die vielversprechendsten Modelle mit biologischen Komponenten wie Immunzellen, mechanischen Signalen und spezifischen Gewebeumgebungen.
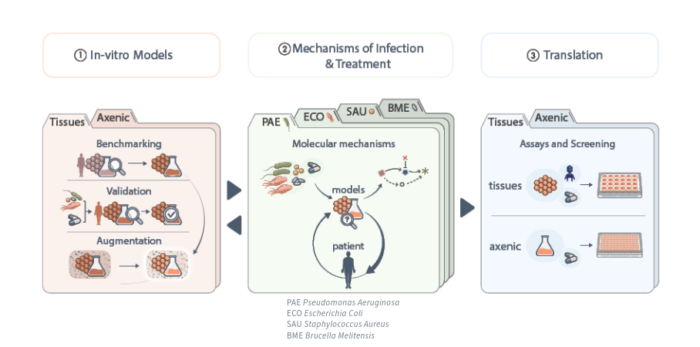
Zweitens nutzen wir diese Modelle, um neue biologische Erkenntnisse zu gewinnen, übersehene Ziele zu identifizieren und kritische Mechanismen von Antibiotika und neuen Hemmstoffen unter physiologisch relevanten Bedingungen zu bestimmen. Daraus ergeben sich mechanistische Hypothesen, die in Kliniken getestet werden können und mit denen klinisch relevante Fragen anhand von In-vitro-Modellen untersucht werden können.
Drittens entwickeln wir auf der Grundlage dieser mechanistischen Arbeit und der Konstruktion verbesserter Modelle für erhöhte Robustheit und Durchsatzleistung Assays für das Screening von Naturstoffen und synthetischen Verbindungen sowie für die Entwicklung unkonventioneller Ansätze.
Diese Arbeit ist in acht Arbeitspakete unterteilt, wie in der obigen Abbildung dargestellt.